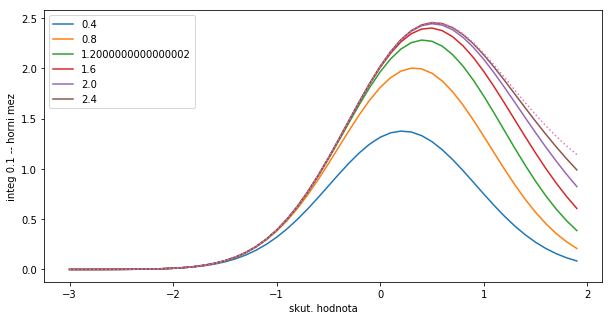
array([ 0.99999999, 0.99999998, 0.99999997, 0.99999996, 0.99999994,
0.99999992, 0.99999988, 0.99999983, 0.99999975, 0.99999963,
0.99999946, 0.99999921, 0.99999885, 0.99999832, 0.99999755,
0.99999642, 0.99999479, 0.99999241, 0.99998895, 0.99998391,
0.99997661, 0.999966 , 0.99995063, 0.99992836, 0.99989615,
0.99984962, 0.99978253, 0.99968594, 0.99954717, 0.99934829,
0.99906404, 0.99865901, 0.99808399, 0.997271 , 0.99612708,
0.99452641, 0.99230091, 0.98922946, 0.98502638, 0.9793306 ,
0.9716977 , 0.96159794, 0.94842445, 0.93151587, 0.91019708,
0.88383912, 0.85193529, 0.81418517, 0.77057294, 0.72142424])